Pattern Classifier
Attenzione: le informazioni in questa sezione riguardano JINGPaC 1.0
Per informazioni relative alla versione di JINGPaC più aggiornata (v2.0) fare riferimento
a questa pagina .
JINGPaC è composto da tre packages: classifier.gp, classifier.nn e classifier. I primi due sono implementazioni generiche di algoritimi di programmazione genetica e di reti neurali,rispettivamente, sviluppati cercando di astrarli dal problema specifico da affrontare in modo da ottenere una API. L'ultimo è una applicazione che utilizza i metodi definiti dai primi per evolvere un pattern classifier, ovvero ottenere un programma che sia in grado di distinguere, con una buona affidabilità, tra differenti tipi di ingresso e riconoscere la loro appartenenza a un tipo.
La nostra implementazione di JINGPaC si inserisce all'interno di un progetto preesistente il cui scopo è quello di riconoscere delle targhe automobilistiche acquisite tramite una videocamere fissa con un'elevata accuratezza. Perciò il nostro Pattern Classifier è stato progettato per interfacciarsi con i dati in ingresso dati dal sistema, ovvero le singole immagini contenenti un solo carattere già codificato in codice binario.
Uno sguardo approfondito
Durante la fase di evoluzione/addestramente creiamo un classificatore (o un insieme di classificatori) che prenderà come
ingresso i dati da parte del sistema di riconoscimento targhe.
Nel caso dell'approccio tramite programmazione genetica è stato scelto di creare un classificatore per ogni simbolo da
riconoscere, ovvero si ha un classificatore che ha come valore di uscita solo 1 o 0; in particolare esso dovrebbe presentare
1 quando l'input (ovvero l'immagine codificata) corrisponde al tipo di classificatore. In questo modo se si vuole riconoscere
un'immagine questa dovrà essere valutata da tutti i classificatori (uno per ogni simbolo) creati tramite programmazione genetica.
Un problema di questo approccio è quello di avere più di uno (o nessun) classificatore con uscita alta a seguito di un determinato
input, per questo motivo abbiamo introdotto anche un classificatore basato su reti neurali che permette una classificazione
univoca del dato in ingresso, anche se a spese delle prestazioni, che verrà utilizzato solo nei casi non riconosciuti tramite
i classificatori generati tramite programmazione genetica.
Infatti in nostro "Function Set" nella programmazione genetica (ovvero le funzioni che possono essere utilizzate per creare i
programmi) è composto da soli operatori bitwise (ovvero che lavorano bit a bit) in modo da mantenere molto elevate le prestazioni
di velocità in fase di esecuzione. Di conseguenza il "Terminal Set" è composto dai singoli bit dell'ingresso per così avere un programma
totalmente bitwise.
Il sistema di riconoscimento targhe può fornire in ingresso due tipi di dati: uno esteso e uno ridotto. Nel formato ridotto
i primi 28 caratteri corrispondo a 28 bit che a loro volta sono una codifica dell'immagine mentre il 28° carattere corrisponde
al simbolo/numero atteso. Nel formato esteso la stringa di input è data da quattro numeri long e il quinto numero corrisponde all'output
atteso.
0110100110011001100111110110 0
0011001001100100111111110010 4
1110100111111001100111100000 8
Esempio di input esteso:
15171080 12829635 12829635 1014940611 0
982268 16776974 12615810 3993539 5
6503936 12829507 12647670 1014941376 9
Diagramma UML del package classifier:

"Classifier" è l'interfaccia principale dell'applicazione che viene implementata da "NNClassifier" e "GPClassifier" che
sono, rispettivamente, le classi astratte più alte del classificatore funzionante tramite reti neurali e programmazione genetica.
A loro volta queste classi sono estese da "NNSuperVC" e "GPSuperVC" che astraggono un classificatore supervisionato; è perciò possibile
creare un classificatore non supervisionato aggiungendo qui le relative classi astratte. Infine "BackPropagationSVC" e "GPEvolutionSVC"
sono le classi concrete che devono essere istanziate per generare ogni classificatore.
L'interfaccia "TrainingSet" si occupa di fornire un metodo comune di accesso ai dati di input del sistema che devono essere caricati
attraverso un'apposita implementazione dell'interfaccia "DataReader".
Infine "TrainReport" è l'interfaccia che gestisce le statistiche.
Genetic Programming
Attenzione: le informazioni in questa sezione riguardano JINGPaC 1.0
Per informazioni relative alla versione di JINGPaC più aggiornata (v2.0) fare riferimento
a questa pagina .
Il package classifier.gp è la parte di JINGPaC relativa alla programmazione genetica e contiene
le interfaccie, le classi astratte e le relative implementazioni di quelle classi che implementano funzioni indipendenti
dal tipo di problema utilizzato come, ad esempio, le funzioni di selezione degli individui, la struttura dati ad albero, gli operatori
genetici come il crossover e la mutazione. Inoltre sono presenti le classi astratte che devono essere implementate diversamente
in base al problema che deve essere analizzato come la funzione di fitness, il function set, il terminal set.
Se è necessario avere maggiori chiarimenti sulla programmazione genetica in generale è meglio passare alla
sezione link prima di proseguire.
Analizziamo ora le parti principali della parte dell'api riguardante la programmazione genetica.
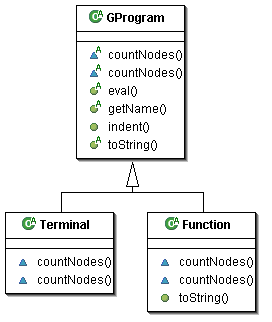
Abbiamo deciso di utilizzare una rappresentazione ad albero dei programmi perciò la classe astratta "GProgram"
definisce i possibili tipi di nodi di cui può essere composto il nostro programma. I nodi terminali devono contenere un valore
ed sono le foglie dell'albero, mentre i nodi funzione sono i nodi dell'albero da cui partono uno o più rami.
Nell'applicazione del pattern classifier abbiamo utilizzato come terminal set i singoli bit di input e come function set solo funzioni
bitwise (v. Pattern Classifier).
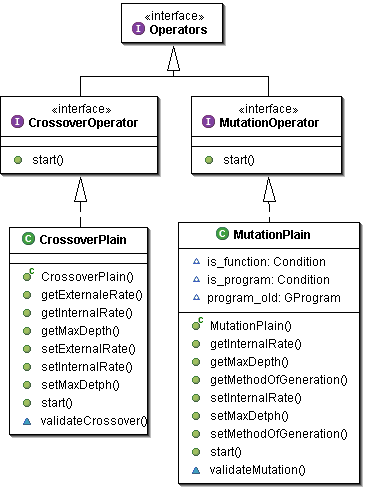
Gli operatori genetici ricoprono un ruolo di fondamentale importanza visto che è affidato ad essi il compito selezionare all'interno
dell'individuo (e quindi dell'albero) quale parte mutare o scambiare con un altro individuo. È; fornita sia l'interfaccia generica
per ogni operatore, sia quella specifica per ogni tipo di operatore inoltre è stato fornita una implementazione delle stesse.
Gli operatori necessitano di alcuni parametri per operare come, ad esempio, i possibili criteri di selezione del nodo dell'albero da scegliere.
Nella nostra implementazione quando l'operatore di mutazione sceglie un nodo genera un nuovo albero che sostituisce il sottoalbero presente
a partire da quel nodo (salvo rispettare vincoli come quello della profondità massima); analogamente l'operatore di crossover scambia i
due sottoalberi sottostanti ai nodi scelti tra i due individui.
E' possibile perciò aggiugere nuove implementazioni degli operatori genetici semplicemente implementado la relativa interfaccia.
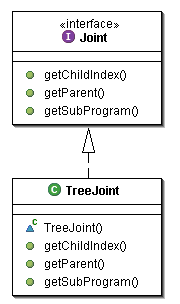
Questa è la struttura dati che permette agli operatori genetici di operare, infatti permette di immagazzinare tutte le informazioni contenute
in un nodo
La funzione di fitness misura il grado di "bontà" di un determinato programma ed è la stima utilizzata durante la scelta
degli individui da sottoporre agli operatori genetici. Per questo motivo è di fondamentale importanza che essa sia
equilibrata in modo da dirigere non l'evoluzione verso uno stallo. Il problema principale riguardo a ciò è che non
esiste la "ricetta" perfetta e inoltre ogni funzione di fitness è altamente specifica al problema che si sta trattando.
Nella applicazione del pattern classifier abbiamo implementato più funzioni di fitness tra cui "BinaryClassifierFitness"
che è basata sull'accuratezza dei programmi testati; inolre abbiamo cercato di ottenere un classificatore che avesse
il numero più basso di falsi negativi (ovvero quando l'output è 0 mentre dovrebbe essere 1, ovvero non ha riconosciuto un
pattern del suo stesso tipo) mantenendo comunque valori molto bassi di falsi positivi (ovvero se presenta un valore di uscita
1 quando l'input non è relativo allo stesso pattern). Questo perchè l'ultimo errore può essere più facilmente recuperato
perchè quando l'uscita di più classificatori è a 1 è possibile utilizzare la rete neurale. Perciò abbbiamo pesato l'importanza
dei falsi negativi ddi un fattore equivalente al numero dei pattern di diverso tipo su quelli corretti:
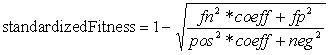
dove:
fn = falsi negativi
fp = falsi positivi
pos = occorrenze dello stesso pattern del classificatore nel training set
neg = occorrenze dei pattern diversi dal classificatore nel training set
coeff = neg / pos

La funzione di selezione viene utilizzata per selezionare gli individui da passare agli operatori genetici, utilizza il valore
della fitness di ogni individuo come stima della bontà dello stesso.
Nell'applicazione del pattern classifier abbiamo creato varie implementazioni dell'interfaccia "SelectionFuncition" ma è possibile
crearne di altre semplicemente aggiungendo. Ad esempio:
"WorstSelection" seleziona l'individuo con la fitness più bassa;
"BestSelection" seleziona l'individuo con la fitness più alta;
"FitnessProportionate" seleziona casualmente in modo proporzionale alla fitness (chi ha una fitness più elevata ha maggiori
probabilità di essere selezionato),
"FitnessOverselected" utilizza 2 parametri (CUTOFF e PROPORTION). Partiziona la popolazione in due gruppi in base alla fitness
di dimensioni definite dal valore CUTOFF; successivamente sceglie il gruppo casualmente in base a PROPORTION e dentro di esso
seleziona gli individui tramite FitnessProportionate.
GPEvolution viene chiamato da GPEvolutionSVC e gestisce l'avvicendarsi delle generazioni e degli scambi degli individui tra popolazioni
multiple.
Breeder: è la classe utilizzata per generare i nuovi individui durante la fase riproduttiva di ogni generazione
Individual: è una struttura dati utilizzata per memorizzare, il programma, la sua fitness ed altri dati relativi alle sue prestazioni.
Population: contiene tutti gli individui all'interno della popolazione, possiede metodi per generare nuovi individui e per inizializzare
la popolazione stessa. Inoltre gestisce anche l'evoluzione della popolazione per la generazione corrente.
Un problema fondamentale quando si sviluppa un programma genetico su una macchina deterministica (come sono gli odierni pc) è quello
di ottenere numeri casuali "buoni". Ovvero essendo i computer macchine deterministiche è difficile ottenere numeri realmente casuali: per
questo motivo abbiamo generato una classe che fornisca numeri casuali a tutti i metodi che ne richiedono senza quindi rischiare di avere
numeri casuali generati dallo stesso seme o comunque simili.

Nonostante JINGPaC sia una API, e perciò è possibile impostare tutti i parametri tramite chiamate a funzione, abbiamo implementato anche
un metodo di passaggio dei parametri tramite file di testo. La classe Parameters si preoccupa di leggere da questo file di testo tutte
le opzioni settate e di completarle con i parametri standard presenti nella classe stessa.
num_of_population: 1 //numero di popolazioni
exch_cycle: 0 //ogni quanti cicli evolutivi avviene lo scambio di individui tra le popolazioni
num_of_exchange: 0 //numero di scambi da effettuare
max_generation: 500 //numero massimo di generazioni dopo il quale ha termine l'evoluzione se non viene raggiunto il limite impostato
//nel caso di popolazioni multiple occorre indicare anche il numero della popolazione; es: population[0].size: 200 o population[1].size=500
population[0].elitism: false //abilita l'elitismo (l'individuo migliore di una generazione viene copiato in quella successiva
population[0].size: 300 //dimensione della popolazione
population[0].depth: 2-6 //profondità degli alberi quando vengono generati
population[0].random_attempts: 100 //se vengono generati due individui uguali cercherà di generarne uno diverso per il numero indicato di volte, oltre il quale lo terrà uguale
population[0].method_of_generation: 3 //metodo di generazione dell'albero: FULL = 1; GROW = 2; RAMPED_HALF_AND_HALF = 3
population[0].breeder.max_depth: 17 //dimensione massima di un albero
population[0].breeder.crossover_rate: 0.7 //probabilità che venga scelto il crossover come operatore
population[0].breeder.mutation_rate: 0.2 //probabilità che venga scelta la mutazione come operatore
population[0].breeder.reproduction_rate: 0.1 //probabilità che venga scelto la riproduzione come operatore
//IMP: La somma di questi tre valori deve risultare 1
//I metodi di selezione sono: TOURNAMENT = 1; FITNESSOVERSELECTED = 2; FITNESSPROPORTIONATE = 3; RANDOMSELECTION = 4; BESTSELECTION = 5; WORSTSELECTION = 6;
population[0].breeder.crossover.select: 1 //metodo di selezione per il crossover per il primo individuo
population[0].breeder.crossover.select.tournament.size: 7 //dimensione del gruppo per la scelta del primo individuo
population[0].breeder.crossover.select.fitness_overselected.proportion: 0.80 //parametro per la scelta del primo individuo
population[0].breeder.crossover.select.fitness_overselected.cutoff: 0.32 //parametro per la scelta del primo individuo
population[0].breeder.crossover.select2: 1//metodo di selezione per il crossover per il secondo individuo
population[0].breeder.crossover.select2.tournament.size: 7 //dimensione del gruppo per la scelta del primo individuo
population[0].breeder.crossover.select2.fitness_overselected.proportion: 0.80 //parametro per la scelta del secondo individuo
population[0].breeder.crossover.select2.fitness_overselected.cutoff: 0.32 //parametro per la scelta del secondo individuo
population[0].breeder.crossover.keep_trying: false //nel caso di generazione di individui oltre le dimensioni massime sceglie se ripetere il crossover o se interrompere
population[0].breeder.crossover.internal: 0.9//probabilità che l'operatore crossover selezioni un nodo funzione come punto di rottura
population[0].breeder.crossover.external: 0.1//probabilità che l'operatore crossover selezioni un nodo terminale come punto di rottura
population[0].breeder.reproduction.select: 5//metodo di selezione per l'operatore di riproduzione
population[0].breeder.reproduction.select.tournament.size: 7//dimensione del gruppo per la scelta dell'individuo
population[0].breeder.reproduction.select.fitness_overselected.proportion: 0.80//parametro per la scelta dell'individuo
population[0].breeder.reproduction.select.fitness_overselected.cutoff: 0.32//parametro per la scelta dell'individuo
population[0].breeder.mutation.select: 3//metodo di selezione per l'operatore di mutazione
population[0].breeder.mutation.select.tournament.size: 7//dimensione del gruppo per la scelta dell'individuo
population[0].breeder.mutation.select.fitness_overselected.proportion: 0.80//parametro per la scelta dell'individuo
population[0].breeder.mutation.select.fitness_overselected.cutoff: 0.32//parametro per la scelta dell'individuo
population[0].breeder.mutation.internal: 0.9//probabilità che l'operatore selezioni un nodo funzione come punto di mutazione
population[0].breeder.mutation.external: 0.1//probabilità che l'operatore selezioni un nodo funzione come punto di mutazione
population[0].breeder.mutation.method_of_generation: 2//metodo di generazione dei sottoalberi mutati (v. crossover)
population[0].breeder.mutation.depth: 17//profondità massima degli alberi mutati
//bisogna indicare tra [] il numero di scambio relativo alle impostazioni; il numero massimo è impostato nel paramentro "num_of_exchange"
exch[0].to: 0//numero della popolazione che riceve gli individui
exch[0].count: 0//numero degli individui da scambiare
exch[0].from: 0//numero della popolazione che riceve gli individui
exch[0].to_select: 1//metodo per selezionare gli individui da mandare
exch[0].from_select: 2//metodo per selezionare gli individui da eliminare per accogliere i nuovi
exch[0].from_select.tournament.size: 7//dimensione del gruppo per la scelta dell'individuo da mandare
exch[0].from_select.fitness_overselected.proportion: 0.80///parametro per la scelta dell'individuo da mandare
exch[0].from_select.fitness_overselected.cutoff: 0.32///parametro per la scelta dell'individuo da mandare
exch[0].to_select.tournament.size: 7//dimensione del gruppo per la scelta dell'individuo
exch[0].to_select.fitness_overselected.proportion: 0.80//parametro per la scelta dell'individuo da eliminare
exch[0].to_select.fitness_overselected.cutoff: 0.32//parametro per la scelta dell'individuo da eliminare
È possibile impostare tutti questi parametri tramite una comoda interfaccia grafica: ParamGUI sviluppata appositamente per questo scopo.
Neural Network
Attenzione: le informazioni in questa sezione riguardano JINGPaC 1.0
Per informazioni relative alla versione di JINGPaC più aggiornata (v2.0) fare riferimento
a questa pagina .
Il package classifier.nn contiene le classi di struttura per la creazione di Network per il loro attraversamento e algoritmi di addestramento per reti neurali.
NetworkComponent
Una generica rete è formata da 5 componenti; 3 classi di struttura (Network, Layer , Node) e due interfacce le cui implementazioni inizializzano la rete secondo una data topologia (NetworkMaker) e ne definiscono l'algoritmo di attraversamento (NetworkEngine).
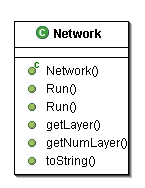
Network
L'implementazione di una rete passa attraverso la creazione di un oggetto Network.
Un oggetto Network incapsula un oggetto NetworkEngine ed un oggetto NetworkMaker, oltre ad oggetti Layer.
Il costruttore di Network sfrutta l'oggetto che implementa l'interfaccia NetworkMaker per inizializzare i Layer secondo l'opportuna topologia; questa struttura dovrebbe garantire la possibilità di estendere in futuro il package con nuove implementazioni di NetworkMaker ed aumenare così il numero di strutture di rete preimplementate. Allo stadio di sviluppo attuale l'unica implentazione di NetworkMaker è la classe MultiLayerMaker che permette la realizzazione di reti multilivello a percettroni...
L'attraversamento della rete ad opera di un particolare input invece è assicurato dal metodo run(); l'override di tale metodo permette di passare l'input alla rete o come array di Object o attraverso un oggetto che implementi l'interfaccia AbstractList.
Una volta invocato, il metodo run() si appoggia all'omonimo metodo di NetworkEngine la cui implementazione (passata al costruttore di Network) definisce l'algoritmo con cui l'imput deve essere processato attraverso la struttura di rete...; in relazione con quanto suddetto questa architettura permette di definire per ciascuna topologia preimplementata attraverso un oggetto NetworkMaker uno o più algoritmi appropiati di attraversamento. Allo stadio di sviluppo attuale la classe MultiLayerEngine implementa l'algoritmo di Heb per l'attraversamento di una rete multilivello di precettroni; secondo quanto definito da tale algoritmo l'input del nodo i-esimo al livello j-esimo è costituito dalla somma degli output di tutti i nodi al livello j-1-esimo ad esso connessi.
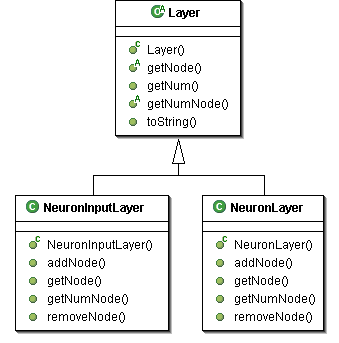
Layer
Un oggetto Layer è fondamentalmente un wrapper di oggetti Node; i metodi associati a questa classe permettono di aggiungere, rimuovere o prelevare un oggetto Node.
Per la realizzazione della rete neurale sono state utilizzate estensioni di questa classe, in particolare, le classi NeuronInputLayer e NeuronLayer che incapsulano rispettivamente oggetti di tipo InputNeuron e Neuron.
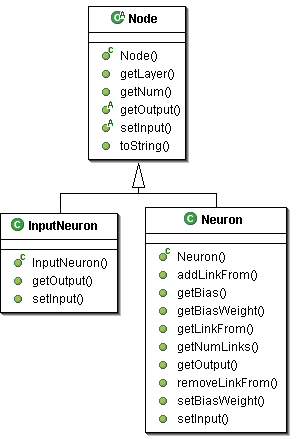
Node, Neuron e InputNeuron
Qualsiasi classe che vada ad estendere la classe astratta Node va a determinare l'unità strutturale più a basso livello della rete; nelle reti neurali tale classe è estesa da Neuron ed InputNeuron.
Ciascun oggetto Neuron è collegato ad altri oggetti Neuron o InputNeuron attraverso oggetti LinkTo e LinkFrom; in questo modo il flusso di dati lungo la rete può essere bidirezionale.
Gli oggetti Link sono gestiti attraverso i metodi di interfaccia pubblici removeLinkFrom() e addLinkFrom() che permettono di aggiungere o rimuovere solo oggetti LinkFrom;la gestione degli oggetti LinkTo infatti è stata resa trasparente al fine di ridurre il pericolo di incoerenza o di illegittimità sulle chiamate ai metodi.
Ogni oggetto Neuron incapsula inoltre un'implementazione di activationFunction; tale oggetto (p.e. un'istanza di Sigmoid) associa al neurone una funzione di attivazione attraverso la quale vengono "filtrati" i dati provenienti dai nodi collegati in ingresso.
Il sistema di attraversamento del'input rispetto al neurone è di tipo "store and forward", ciò significa che invocando il metodo setInput() i dati in ingresso vengono processati dalla funzione di attivazione e quindi bufferizzati nel neurone stesso; il valore di uscita è quindi disponibile invocando il metodo getOutput().
I metodi getBiasWeight() e getBias() ritornano rispettivamente il "peso" della connessione tra il neurone e il "bias" ed il valore di quest'ultimo.
La classe InputNeuron è un semplice buffer per i dati in ingresso alla rete; non è escluso che in stadi più avanzati si sviluppo il suo utilizzo possa rivelarsi ridondante...
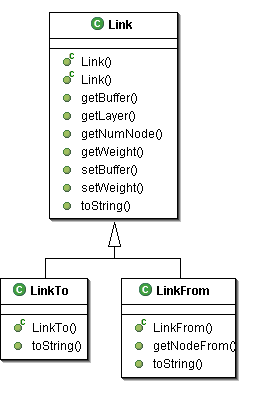
Link,LinkTo,LinkFrom
La classe Link fornisce una struttura di collegamento unidirezionale tra due nodi della rete; ogni nodo pertanto incapsula tanti oggetti LinkTo quanti sono sono i nodi con un collegamento in ingresso e tanti oggetti LinkFrom quanti sono i nodi verso i quali esiste un collegamento in uscita...
Due oggetti LinkFrom e LinkTo operanti tra due stessi nodi condividono inoltre un oggetto Weight ed un oggetto LinkBuffer; il primo misura il "peso" della connessione il secondo serve come buffer per memorizzare qualsiasi valore/oggetto necessario all'applicazione di un particolare algoritmo (p.e. l'oggetto LinkBuffer viene utilizzato nell'algoritmo di retropropagazione dell'errore per memorizzare i valori della delta associati a ciascun Link).
NetworkAlghoritms
BackPropagation
Un'oggetto BackPropagation permette di applicare l'algoritmo di addestramento a retropropagazione dell'errore ad un dato oggetto Network.
In particolare l'addestramento inizia invocando il metodo train(); i parametri di questo metodo sono un oggetto TrainingSetSV ed uno di tipo errorCondition; il primo incapsula un set di addestramento per algoritmi supervisionati, il secondo testa il grado di addestramento della rete e stabilisce una condizione di uscita dall'algoritmo...
I metodi setTestCicle(), setMaxNumOfCicle() permettono di impostare rispettivamente, la frequenza (in numero di cicli) di testing sull'errore espresso dalla rete e il numero massimo di cicli che possono essere effettuati (al raggiungimento dei quali l'addestramento termina anche se non è stata raggiunta la condizione attesa sull'errore).

